在计算生物学领域,一个长期存在的难题终于迎来突破:字节跳动 Seed 团队开源的 Protenix-v1,成为首个在严格对等条件下性能达到甚至超越 AlphaFold 3 的完全开源模型。
这意味着,科研人员、药物开发者和开源社区,终于拥有了一个透明、可复现、高性能的工具,用于预测蛋白质、核酸、配体及其复合物的三维结构——而无需依赖闭源黑箱。
为什么 Protenix 如此重要?
AlphaFold 3 虽然强大,但其闭源性质限制了科学界的深度验证、定制与改进。此前的开源模型(如 Boltz-1、Chai-1)虽有进展,但在关键任务(如抗体-抗原结合预测)上仍存在显著差距。
Protenix-v1 不仅填补了这一空白,更展现出一项独特能力:推理时扩展(Inference-Time Scaling)——通过增加采样数量,预测精度可持续提升,为实际应用提供了明确的“计算成本 vs. 精度”权衡机制。
例如,在抗体-抗原复合物预测中,将采样种子从 5 个增至 80 个,DockQ 成功率从 36% 提升至 47.7%,这是此前开源模型无法实现的。
核心能力:不止于蛋白质
Protenix 支持多种生物分子的通用结构预测:
- ✅ 蛋白质单体与复合物
- ✅ 蛋白质-蛋白质 / 抗体-抗原
- ✅ 蛋白质-小分子配体(药物靶点关键)
- ✅ 蛋白质-RNA / 蛋白质-DNA(调控机制研究)
所有预测均输出 3D 坐标 + 置信度分数(pLDDT、pTM、ipTM),便于后续筛选与实验验证。
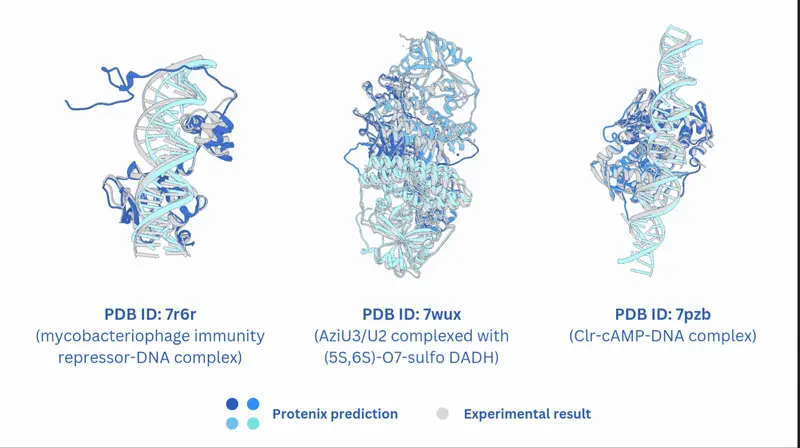
性能表现:全面对标 AlphaFold 3
在修正后的 FoldBench 和自建 PXM 基准集上,Protenix-v1 表现如下:
| 任务类型 | Protenix-v1 | AlphaFold 3 | 优势 |
|---|---|---|---|
| 抗体-抗原 | 52.31 (DockQ SR) | 48.75 | +3.56% |
| 蛋白质-RNA | 68.46 | 65.22 | 显著领先 |
| 蛋白质-配体 | 62.54 | 62.59 | 基本持平 |
| 蛋白质单体 | 0.8857 (lDDT) | 0.8803 | 略优 |
注:所有测试均在相同数据截止日期(2021-09-30)、模型规模与计算预算下进行,确保公平比较。
更令人振奋的是其实用增强版 Protenix-v1-20250630:在引入更新训练数据后,抗体-抗原预测成功率在 PXM-2024 上跃升至 64.02%,展现了强大的可扩展性。
技术亮点
- 推理时扩展行为
增加采样种子数(1 → 80),性能近似对数线性提升,尤其适用于高价值靶点(如新药候选)。 - 增强数据处理
- 整合最多 4 个结构模板
- 支持 RNA MSA(通过 Rfam、RNAcentral)
- 1300 万蒸馏单体结构 + 无序区域专项优化
- 严格评估体系
开源 PXMeter 工具包 与多个高质量基准集(PXM-2024/2025、PXM-Antibody 等),并修正了 FoldBench 中的数据覆盖不一致问题。 - 完整开源生态
不仅开源模型,还包含:- PXDesign:蛋白质-结合剂设计框架(实验成功率 20–73%,超现有方法 2–6 倍)
- Protenix-Dock:经典对接工具(无深度学习依赖)
- 训练数据管道、MSA 流程、推理脚本
快速开始
安装与预测极其简单:
# 安装
pip install protenix
# 预测(JSON 输入)
protenix pred -i input.json -o ./output -n protenix_base_default_v1.0.0
项目提供详细 inference_demo.sh 示例,涵盖多分子输入格式、模板使用、置信度过滤等场景。
应用场景
- 药物发现:快速解析靶点结构,指导小分子或抗体设计
- 基础科研:研究蛋白质功能、RNA 调控、病毒入侵机制
- 合成生物学:设计人工酶或代谢通路
- 疫苗开发:精准预测抗原表位
对于高精度需求(如临床前候选),建议启用 80+ 种子采样;对于高通量筛选,标准 5 种子配置即可平衡效率与准确性。
数据统计
相关导航


Confer

DroidRun

nanobot

AgentKit

Project Mariner

微软 Copilot

Antfarm